导语
TargetScan是一款预测miRNA结合位点的软件,对于哺乳动物中miRNA结合位点预测的效果非常好。在预测miRNA靶基因之前,首先需要确定转录本的3’UTR区域,TargetScan数据库通过一种名为3P-seq的测序技术,确定转录本对应的3’UTR区(哺乳动物中的miRNA通过结合转录本序列的3’UTR区,从而发挥转录后调控作用),并且结合该技术的分析结果和NCBI中已有的3’UTR注释,提供一个综合的3’UTR区序列。
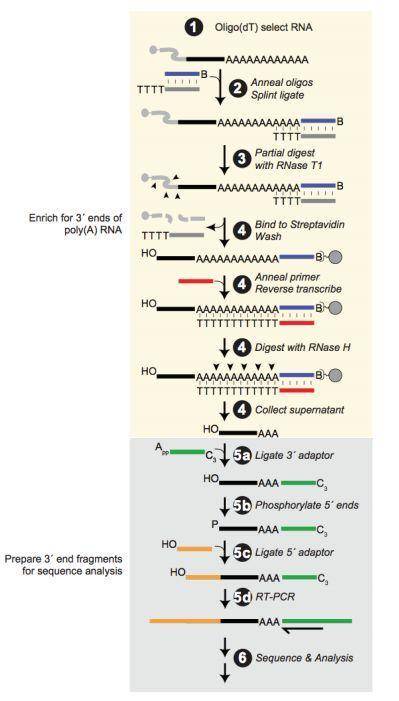 3’UTR区域技术原理示意图
3’UTR区域技术原理示意图
miRNA在不同物种间具有保守性,通过预测不同物种的miRNA靶标位点,targetscan的开发团队发现其结合位点也具有一定的保守性,可以划分成以下几类:
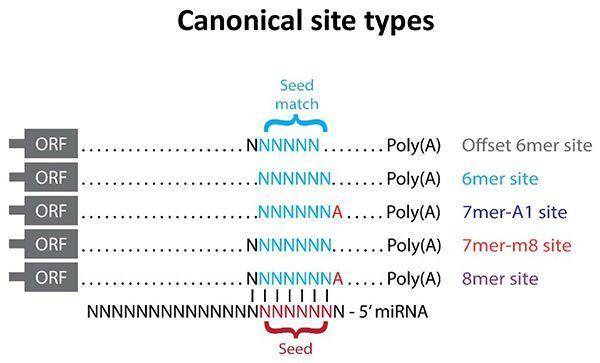
进一步根据结合区域的特定和miRNA的多序列比对结果,划分成不同的miRNA family。
根据序列在不同物种间的保守性,将miRNA family划分成Broadly、poorly、conserved。

TargetScan首页
可以看到,TargetScan主要的功能内容都在首页,快来一探究竟吧:
首先是Search for predicted microRNA targets in mammals,搜索哺乳动物中预测的microRNA靶标 ,可提供的子链接专库有:,
人 : TargetScanHuman
小鼠 :TargetScanMouse
果蝇:TargetScanFly
线虫:TargetScanWorm
斑马鱼 : TargetScanFish
TargetScan搜索功能运用
--输入需要预测的基因名或者输入ENST编号,将显示预测该基因的所有miRNA位点。我们以Hmga2为例,输入好之后,点击Submit即可。

显示内容如下:
Part1

这里是预测结果,有颜色的点,标注了预测靶点。TargetScan7使用3'-UTR配置文件。上面3'-UTR图谱是使用3P-seq标签(在y轴上标记了3'UTR的标签数量)构建的,表明了mRNA切割和聚腺苷酸化位点的位置和用法。将来自多个细胞系或组织的3P-seq标签相互标准化(以说明可变的测序深度),然后汇总到一组共有计数中。将归一化的3P-seq标签分配给每个终止密码子的代表性3'UTR,并将其相加(如配置文件的左侧所示),以量化该终止密码子的用法。这个还包括在Gencode注释的远端添加的5个伪计数。每个代表性3'UTR的注释均以最长的Gencode 3'UTR开始,有时会使用3P-seq或其他注释来源的信息进行扩展。3'-UTR配置文件(红线)随每组群集的3P-seq标签一起下降,指示包含3'UTR片段的转录本比例。每个TargetScan 3'-UTR配置文件还显示Gencode注释(带有Ensembl笔录ID的蓝色垂直线)最远端的位置。每一个预测结果可以点击(每个小颜色方块)。点击后,下方区域信息会更新。
底部显示的是选中miRNA的靶点周围的序列,左边显示的是其他物种中相应位点的情况。同时,我们能看出miRNA种子序列在UTR中的位置,保守的种子序列区会以白色高亮显示。这就是该位点详细的信息,包括miRNA种子区在该区域的位点,位点中间的序列,位点的种类及位点评分信息。context++score评分越低,位点是靶点的概率越大,此外,percentile就是score的换算,数值越接近100,位点是真正靶点的概率越大。

Part2
Conserved。这一部分会显示位置与Predicted consequential pairing of target region (top) and miRNA (bottom)预测的目标区域(顶部)和miRNA(底部)的配对情况、类型、上下文++得分及其百分比等内容。
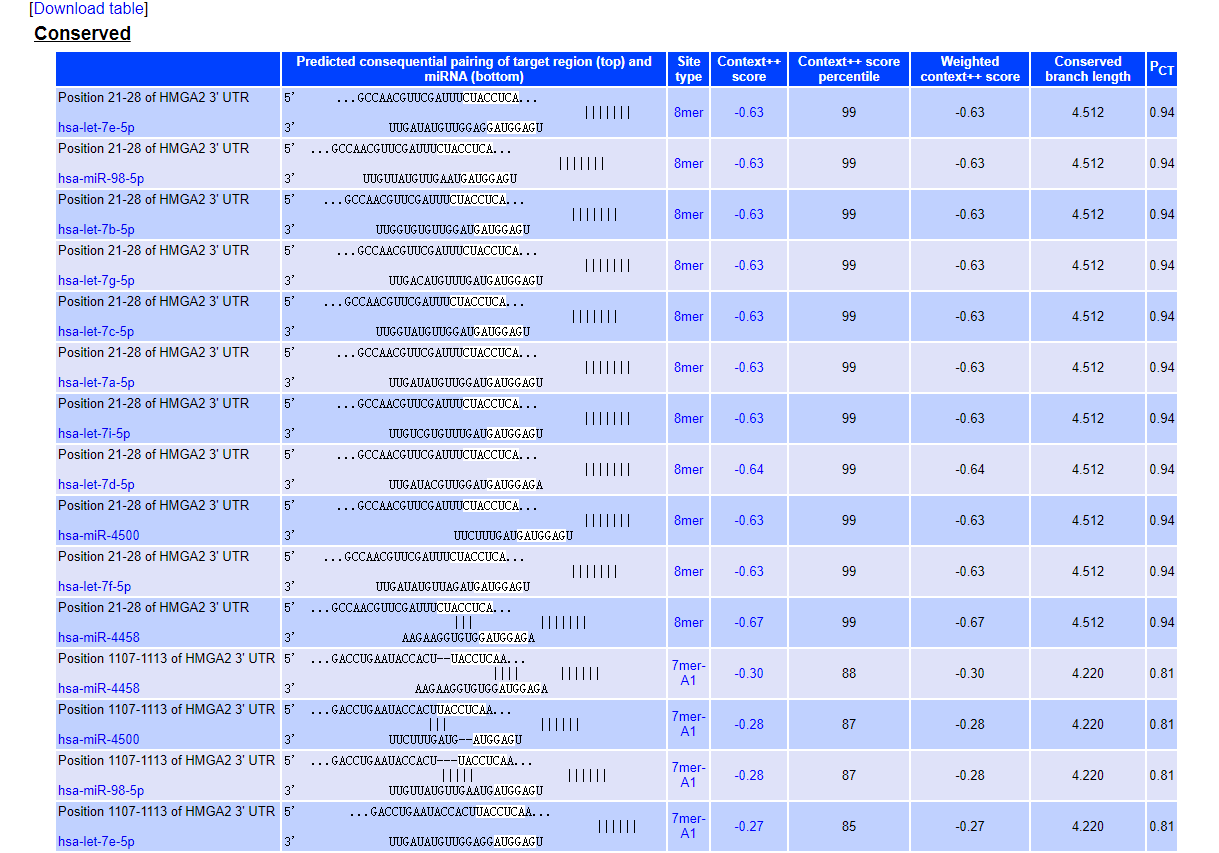
这个表格如果想下载也是没有问题的。点击Site type,呈现结果:
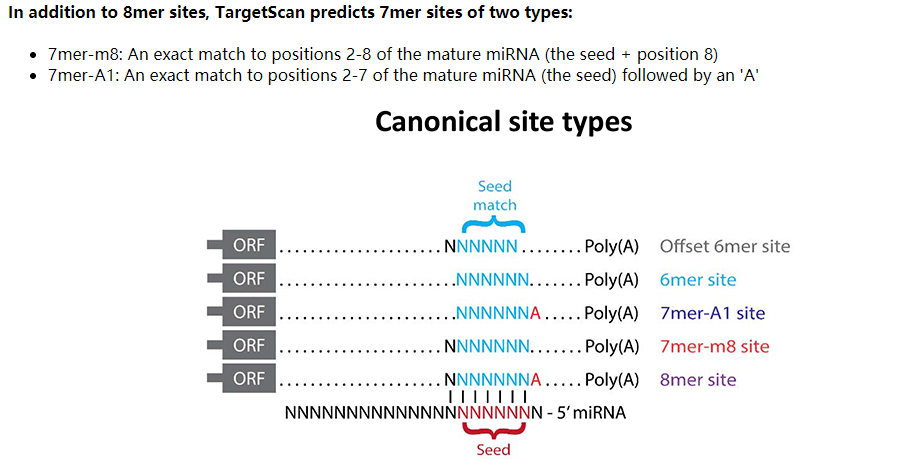
7mer-m8:与成熟miRNA的2-8位(种子+ 8位)完全匹配
7mer-A1:与成熟miRNA(种子)的2-7位完全匹配,后跟“ A”
点击Context++ score 会出现14个打分功能维度:

如果回到点击View table of miRNA sites,将显示PTEN的UTR上的可能的所有miRNA。点击任一miRNA后,回到UTR页面。最后两列是每个miRNA总体评分情况:total context++score代表所有的这个miRNA的评分加和。加和越小,miRNA以这个蛋白为靶点的概率越高。aggregate Pct代表的是总体的miRNA位点的保守情况。
以上就是TargetScan预测miRNA靶基因的主要攻略啦,如果写文章使用了该数据库,可以对以下文献进行引用:To reference information from this database, please cite one of the following papers:Agarwal V, Bell GW, Nam J, Bartel DP. Predicting effective microRNA target sites in mammalian mRNAs. eLife, 4:e05005, (2015)返回搜狐,查看更多
责任编辑: